헬스케어와 생명과학을 위한 AWS 기반 AI 유전체 분석 자동화 가이드
서론
생물정보학과 정밀의학의 발전에 따라, 유전체 분석의 효율성과 속도는 임상과 연구 현장의 핵심 요소로 자리 잡고 있습니다. 그러나 수백만 개의 변이 데이터를 처리하고 임상적으로 유의미한 정보로 변환하기 위해서는 고성능 컴퓨팅 자원과 복잡한 파이프라인이 필요하며, 이를 운영하기 위한 기술 인력이 필수적입니다.
본 글에서는 AWS HealthOmics, Amazon S3 Tables, 그리고 Amazon Bedrock AgentCore 위에서 동작하는 Strands Agent를 활용하여 유전체 변이 해석 파이프라인을 자동화하고, 자연어 인터페이스를 통해 임상 연구자가 직접 질의하고 해석할 수 있도록 돕는 방법과 활용 사례를 소개합니다.
본론
- 주요 구성요소 및 작동 방식
AWS에서 제공하는 AI 기반 유전체 분석 플랫폼은 다음과 같은 주요 요소로 구성됩니다:
- AWS HealthOmics: VCF 파일을 자동으로 VEP(Variant Effect Predictor)를 통해 주석화
- Amazon S3 Tables: PyIceberg를 적용하여 VCF 데이터셋을 컬럼 기반 구조 테이블로 변환
- Amazon Bedrock AgentCore: LLM 기반의 자연어 인터페이스 제공
- Strands Agent SDK: 유전체 질의에 최적화된 에이전트 설계 및 배포 지원
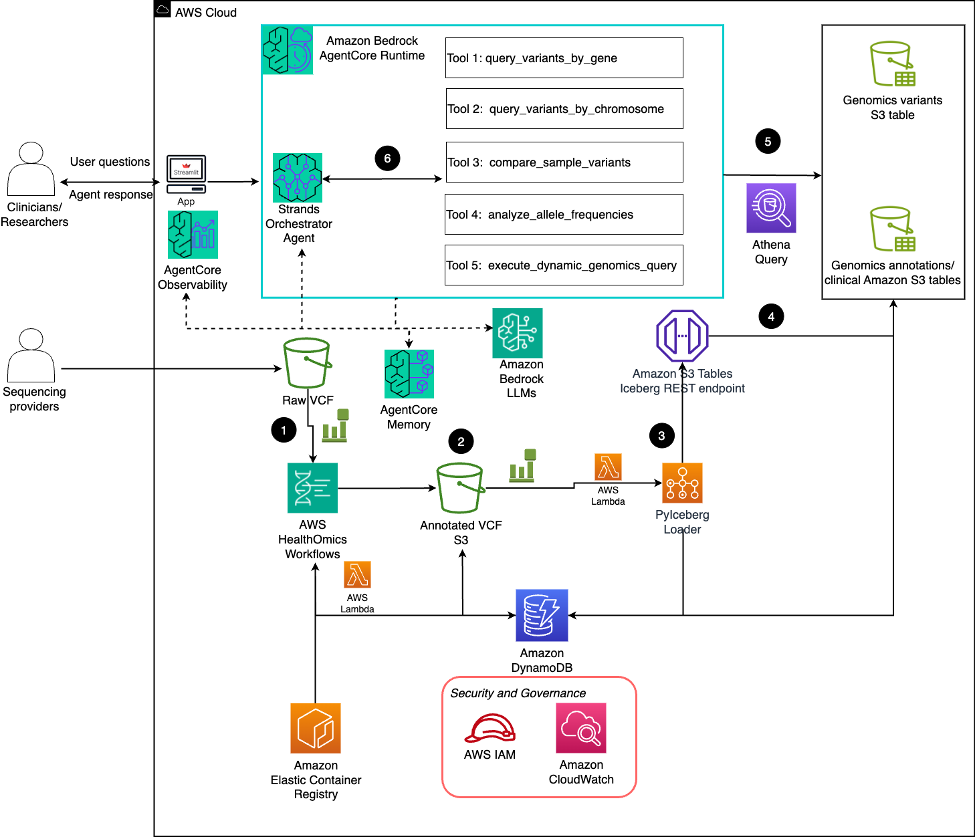
전체 데이터 흐름은 다음과 같이 처리됩니다:
-
원시 VCF 파일을 Amazon S3에 업로드하면, AWS Lambda가 트리거되어 HealthOmics 워크플로우 실행
-
HealthOmics 워크플로우는 VEP을 사용해 기능적 및 임상적 주석을 자동화
-
Amazon EventBridge가 주석화 완료 이벤트를 감지해 다음 단계로 넘김
-
PyIceberg 모듈이 데이터 정제 및 S3 Table 구조에 적합하게 변환하고 Glue Data Catalog에 등록
-
Amazon Athena를 통해 SQL 질의 분석 가능
-
Strands Agent를 통해 자연어 기반 쿼리 실행
-
다양한 자동화 질의 시나리오
에이전트를 통해 수행 가능한 대표적인 활용 시나리오는 다음과 같습니다:
- "BRCA1 유전자에 병원성 변이가 있는 환자를 알려줘"
- "이 코호트에서 statin 저항성 변이가 가장 많은 환자는 누구인가?"
- "환자 NA21144에 대한 위험도 분석 및 임상적 의미 요약해줘"
이러한 질의는 AgentCore의 자연어 인터페이스와 Athena SQL 변환 모듈을 통해 자동 수행되며 결과는 테이블 형식으로 시각화됩니다.
- 고성능 데이터 처리 및 자동화 이점
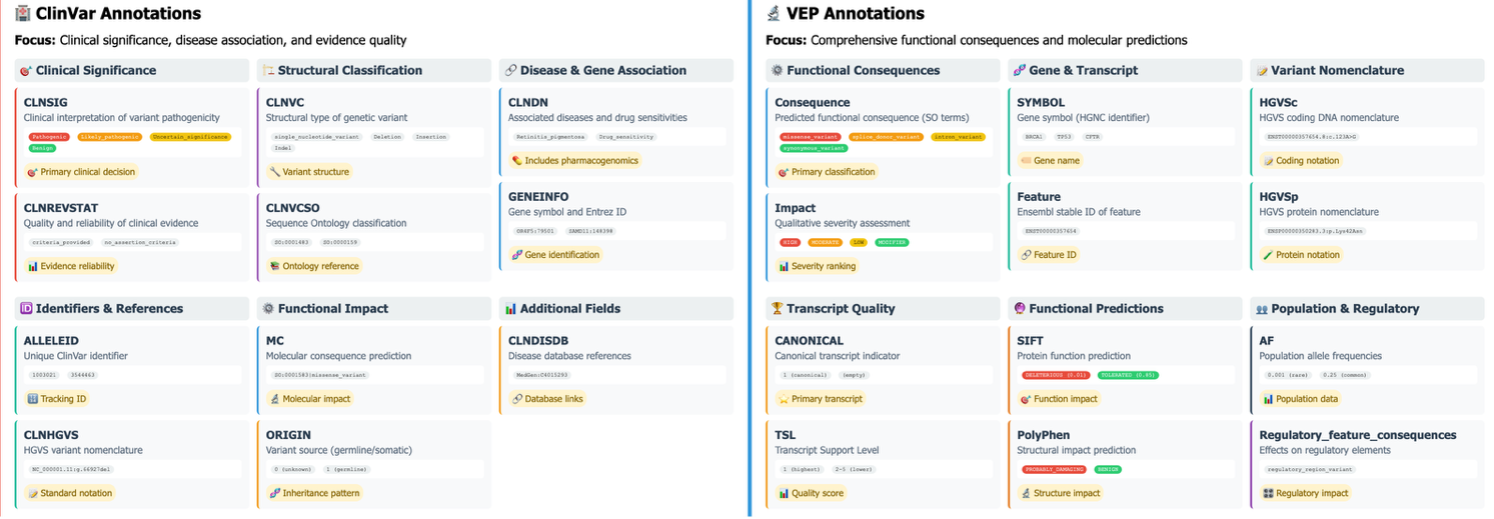
Amazon S3 Tables와 PyIceberg는 모든 VEP 및 ClinVar 주석 데이터를 컬럼 구조 기반 Iceberg 형식 테이블로 저장하며, 이를 통해 다음과 같은 분석 최적화를 실현합니다:
- 수백만 개 변이 데이터를 빠르게 필터링 및 탐색
- 코호트 간 비교 분석 (e.g. 알렐 빈도, 변이 유형별 분포 등)
- 자연어 기반 타겟 질의 변환 (e.g. "chr10 특정 위치 변이 있는 환자 전체 보여줘")
이로써, 기존에는 많은 시간이 소비되던 SQL 스크립트 개발이 필요 없이 바로 임상적 통찰을 얻을 수 있습니다.
- 복잡한 질의도 해결하는 자연어 인터페이스와 사용성
Strands Agent는 다음과 같은 고급 쿼리도 자연어로 받아 처리할 수 있습니다:
- 인구 유전학 분석 (e.g. "코호트와 1000젠 데이터셋 간 병원성 변이의 알렐 빈도 차이 알려줘")
- 병용 변이 위험도 예측 (e.g. "ADRA2A 유전자 변이를 가진 환자 중 인슐린 저항성 연관 고위험 변이도 함께 있는 환자를 알려줘")
- 임상 결정 지원 (e.g. "이 환자의 약물 반응 유전자 변이 기반 추천 용량 전략 알려줘")
이러한 워크플로우를 통해 기존 며칠이 걸리던 복잡한 분석도 수 분 내 결과를 확보할 수 있습니다.
- 자동화된 분석의 한계와 고려 사항
본 자동화 구조는 Lambda 시간 제한(15분), 코호트 규모에 따른 Iceberg 스키마 최적화 난이도 등의 한계도 존재합니다. 따라서 대규모 GVCF 파일의 경우 AWS ECS 또는 Batch 환경 활용 권장되며, 분석 목적에 따른 스키마 구조의 분리도 고려해야 합니다.
결론
AWS의 AI 기반 유전체 변이 해석 자동화 솔루션은 복잡한 생물정보학 파이프라인을 손쉽게 배포하고, 자연어 인터페이스로 접근성 높은 AI 분석 환경을 제공합니다.
특히 AWS HealthOmics, Amazon S3 Tables 기반의 Iceberg 변환, Strands Agent와 Amazon Bedrock AgentCore를 통한 질의 수행은 기존 연구자들이 갖고 있는 분석 역량의 한계를 넘어 자동화와 효율화된 플랫폼을 경험할 수 있게 합니다. 유전 질환 진단, 약물 반응 분석, 인구 기반 리스크 비교 분석까지 다양한 활용 사례가 가능한 이 솔루션은 정밀의학과 바이오헬스케어 도입을 가속화하고 있습니다.
해당 솔루션은 오픈소스로 제공되며, 다음 GitHub 저장소에서 사용 예제를 참조해 직접 활용할 수 있습니다.
AI, Cloud 관련한 문의는 아래 연락처로 연락주세요!
(주)에이클라우드
이메일 : acloud@a-cloud.co.kr
회사 번호 : 02-538-3988
회사 홈페이지 : https://www.a-cloud.co.kr/
문의하기
